Metody izolace DNA
A) Izolace DNA z čerstvého nebo sušeného rostlinného materiálu s použitím Invisorb Spin Plant Mini Kit (INVITEK)B) Extrakce DNA ze sušeného rostlinného materiálu – CTAB metoda
C) Extrakce DNA pomocí NaOH
D) Měření koncentrace DNA a ředění DNA
E) Otestování kvality DNA na agarosovém gelu
A) Izolace DNA z čerstvého nebo sušeného rostlinného materiálu s použitím Invisorb Spin Plant Mini Kit (INVITEK)
Princip
Metoda využívá unikátní patentované technologie Invisorb®. Umožňuje rychlou a účinnou izolaci kvalitní genomické DNA z čerstvého, mraženého nebo suchého rostlinného materiálu (např. z listů, kořenů, plodů nebo semen), také z řas a sinic.
Podstatou je interakce mezi negativně nabitými fosfátovými skupinami DNA a pozitivně nabitými skupinami na povrchu membrány v kolonce, kde se DNA váže a po přečištění se uvolní vhodným elučním pufrem. Vazba nebo naopak uvolnění DNA z kolonky závisí na koncentraci solí a pH použitých pufrů.
Postup
Obecné
- Před použitím promývacích pufrů (Wash Buffer I a II) zkontrolujeme přidání ethanolu (zaškrnutí příslušného políčka na láhvi).
- K času pro centrifugaci přičítáme čas nutný k dosažení požadovaných otáček.
- Odpadní filtráty vzniklé během izolace vyléváme do připravené kádinky.
- Proteinasa K je součástí kitu. Dodává se se v lyofilizovaném stavu. Před prvním použitím přidáme 1 ml sterilní vody a rozpustíme převracením zkumavky v ruce, krátce stočíme.
- Před vlastní prací odpipetujeme do 1,5 ml eppendorfky potřebné množství elučního pufru (Ellution buffer D) a předehřejeme v termobloku na 65°C.
Homogenizace
- V tekutém dusíku vymrazíme 1,5 ml eppendorfku s vloženým rostlinným materiálem (cca 60 mg rostlinné tkáně v čerstvém stavu nebo odpovídající množství sušeného materiálu). Vymrazíme homogenizátorek a krouživými pohyby drtíme vzorek ve vymražené eppendorfce.
- Jinou možností je drtit vzorek ve vymražené třecí misce. Rozdrcený materiál vymraženým skalpelem převedeme do připravené eppendorfky.
- K drcení vzorku je možné použít také mořský písek.
- V některých případech je možné drtit přímo čerstvý nebo suchý materiál za pokojové teploty přímo v lyzačním pufru (Lysis buffer P) bez použití kapalného dusíku.
Lýza
- K rozdrcenému materiálu přidáme 400 µl lyzačního pufru (Lysis buffer P) a 20 µl proteinázy K (rozštěpí proteinů, inaktivace DNas). Lyzační pufr a proteinasu přidáváme dříve než vzorek zcela rozmrzne.
- Vortexujeme a necháme 30 min nebo déle inkubovat při 65°C. Během inkubace 2–3× promícháme.
- Připravíme si kolonky (Spin Filter) do 2,0 ml sběrných zkumavek (Receiver Tube).
Filtrace
- Lyzační směs přeneseme na kolonku (nejen roztok, ale i rozdrcenou tkáň). Centrifugujeme 1 min při 12 000 rpm (dle potřeby déle). Kolonky vyhodíme.
Odstranění RNA
- Pokud je potřeba pro další analýzy odstranit RNA, přidáme 5–40 µl RNAasy A (firma Promega; 10mg/ml; není součástí kitu). Množství závisí na typu a výchozí navážce materiálu.
- Vortexujeme a necháme 5 min inkubovat při pokojové teplotě.
Navázání DNA
- Přidáme 200 µl Binding Buffer P a vortexujeme.
- Umístíme nové kolonky do 2,0 ml sběrných zkumavek. Suspenzi přeneseme na kolonku a inkubujeme 1 min.
- Centrifugujeme 1 min při 12 000 rpm.
Promytí I
- Odstraníme filtrát a kolonky umístíme zpět do 2,0 ml sběrných zkumavek.
- Přidáme 550 µl promývacího pufru I (Wash Buffer I) a centrifugujeme 1 min při 12 000 rpm.
- Odstraníme filtrát a kolonku umístíme zpět do 2,0 ml sběrné zkumavky.
Promytí II
- Přidáme 550 µl promývacího pufru II (Wash Buffer II) a centrifugujeme 1 min při 12 000 rpm.
- Odstraníme filtrát a kolonku umístíme zpět do 2,0 ml sběrné zkumavky.
- Tento krok opakujeme ještě jednou.
Vysušení
- Pro odstranění zbytků ethanolu z promývacího pufru centrifugujeme 2 min při 12 000 rpm.
Eluce
- Kolonky umístíme do sterilních 1,5 ml eppendorfek (nejsou součástí kitu). Pokud předpokládáme delší skladování DNA, použijeme tzv. safe-lock eppendorfky.
- Přidáme 50–100 µl elučního pufru (Elution Buffer D) předehřátého na 65°C. Inkubujeme 10–25 min při pokojové teplotě v uzavřené digestoři bez odtahu.
- Centrifugujeme 1 min při 10 000 rpm.
- Pro získání maximálního výtěžku DNA je možné eluovat nejprve 50 µl elučního pufru a v druhém kroku dalšími 50 µl elučního pufru. Pokud nám nejde o celkový výtěžek DNA, ale o co nejvyšší koncentraci DNA, pak k eluci v druhém kroku použijeme první eluát (tj. roztok DNA pipetujeme znovu na kolonku). Inkubujeme 5 - 10 min a centrifugujeme.
Pozn.: Při centrifugaci nelze zavřít víčka. Při vkládání eppendorfek do centrifugy směrujeme otevřená víčka ve směru otáčení rotoru, abychom zabránili ulámání víček během centrifugace.
B) Extrakce DNA ze sušeného rostlinného materiálu – CTAB metoda
Princip
Metoda je založena na schopnosti CTAB (cetyltrimetylamoniumbromid) vytvářet komplex s nukleovými kyselinami, který je při vysoké koncentraci solí rozpustný (0,7M NaCl), ale při snížené koncentraci solí (0,45M NaCl) vytváří sraženinu. CTAB zároveň působí jako detergent, který uvolňuje DNA z membrán a proteinů. Na základě rozdílné rozpustnosti CTAB v porovnání s DNA je lze oddělit a získat dostatečně čistou rostlinnou DNA.
Tuto metodu lze použít k izolaci většího množství DNA.
Postup
Homogenizace
- Viz extrakce DNA kitem Invisorb Spin Plant Mini Kit (INVITEK).
Vlastní extrakce DNA
- K rozdrcenému materiálu přidáme 700 µl roztoku CTAB a 10 µl 2-merkaptoethanolu. Pracujeme v digestoři.
- Zkumavky uzavřeme, krátce promícháme na vortexu a inkubujeme 30 min v termobloku při 60 °C.
- Během prvních minut inkubace přidáme ke každému vzorku „na špičku špachtle“ polyvinylpyrolidonu (PVP).
- Po inkubaci přidáme 500 µl směsi chloroform: isoamylalkohol (24:1). Pracujeme v digestoři
- Dobře uzavřené zkumavky 2–3× převrátíme a necháme cca 5 min stát.
- Centrifugujeme 10 min při 13 800 rpm.
- Supernatant (cca 500 µl) opatrně přepipetujeme do nových označených 1,5 ml eppendorfek. Je třeba dát pozor, abychom pipetovali jen průhledný supernatant. Pracujeme stále v digestoři a použité špičky a ependorfky vyhazujeme do zvláštní nádoby na nebezpečný odpad.
- Přidáme 500 µl vychlazeného isopropanolu (z mrazáku).
- 1–2 × převrátíme (netřepeme) a necháme cca 30 min stát v –20 °C.
- Centrifugujeme 5 min při 13 800 rpm.
- Supernatant opatrně slijeme do kádinky (v digestoři), na dně ependorfky lze vidět drobný matně bílý pelet DNA. Otevřené ependorfky převrátíme dnem nahoru na filtrační papír (buničinu) pro odstranění zbytku kapaliny.
- Přidáme 400 µl vychlazeného 96% ethanolu (z mrazáku).
- Inkubujeme 15 min v termobloku při 37 °C.
- Centrifugujeme 5 min při 13 800 rpm.
- Supernatant opět opatrně slijeme do kádinky (už není nutné v digestoři).
- Přidáme 200 µl vychlazeného 70% ethanolu (z mrazáku) a necháme cca 5 min stát.
- Centrifugujeme 5 min při 13 800 rpm.
- Supernatant opět opatrně slijeme do kádinky a eppendorfky necháme cca 10 - 15 min stát a vyschnout.
- Přibližně 1–2 min vysušíme pelet v otevřených eppendorfkách na termobloku při 37 °C. Sušení ukončíme ve chvíli, kdy lze pelet poklepem (cvrnknutím) v uzavřené eppendorfce oddělit od stěny.
- Vysušený pelet rozpustíme ve 200 µl TE pufru nebo sterilní vody.
- Uzavřené eppendorfky inkubujeme 30 min na termobloku při 37 °C pro úplné rozpuštění.
- Krátce promícháme na vortexu a krátce stočíme na stolní centrifuze.
- DNA uchováváme v ledničce nebo dlouhodobě při –20°C (–80°C).
C) Extrakce DNA pomocí NaOH
Extrakce DNA pomocí NaOH je velmi levnou a rychlou metodou přípravy DNA. Hojně se používaná pro extrakci DNA z mechů.
Princip
Hydroxid sodný naruší buněčné stěny, DNA se uvolní do roztoku a stává se denaturovanou, řetězce jsou separované.
Postup
Obecné
- Takto získanou DNA obvykle není možné dlouhodobě skladovat (degraduje). Lze ji použít pro běžné metody založené na PCR. Pro některé aplikace náročné na kvalitu DNA (např. AFLP) ji použít nelze.
Izolace
- Část nebo celou rostlinku mechu vložíme do eppendorfky, uzavřeme a vymrazíme v tekutém dusíku. Vymrazíme homogenizátorek a krouživými pohyby drtíme vzorek na jemný prášek.
- K rozdrcenému materiálu přidáme 5 µl 0,5M NaOH a drtíme vzorek v roztoku.
- Přidáme dalších 15 µl 0,5M NaOH a pokračujeme v drcení další 1-2 min.
- Centrifugujeme 2 min 13 800 rpm.
- Supernatant přepipetujeme do nové eppendorfky a ředíme 1:10 roztokem 100mM Tris-HCl, pH 8,3. Podle potřeby dál ředíme roztokem 100mM Tris-HCl pufru.
- Skladujeme v mrazáku při –20°C max. 1 měsíc.
D) Měření koncentrace DNA a ředění DNA
Měření koncentrace DNA
- koncentraci DNA měříme pomocí spektrofotometru Biowave II (Biochrom)
- zaznamenáme:
- koncentraci DNA v ng/µl
- poměr A260/A280 – udává znečištění proteiny, u čisté DNA je 1,8 až 2,0
- poměr A260/A230 – pokud je méně než 1,8, značí to přítomnost organických kontaminant – (poly)fenoly, thiokyanáty, sacharidy, močovina apod.; u čisté DNA se pohybuje v rozmezí 1,8-2,0(–2,2)
Ředění DNA
- ředění vypočteme např. podle vzorečku:
- Cpův ............... koncentrace původní (originálního vzorku)
- Cpož ............... koncentrace požadovaná (na kterou chci ředit)
- Vpož ............... objem požadovaný
- Vpip ................ objem pipetovaný (vzorku)
- zř ................... zředění
- Např. chceme 100 µl DNA o koncentraci 30 ng a máme k dispozici DNA o koncentraci 215 ng/µl.
- připravíme 0,8 % agarosový gel
- cca 5 µl nenaředěné DNA smícháme se 2 µl nanášecího pufru LB (loading buffer) a naneseme na gel
- jako žebříček naneseme 6 µl Lambda/HindIII firmy NEB
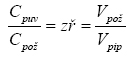

Smícháme tedy 13,95 µl koncentrované DNA (našeho původního originálního vzorku) a 86.05 µl sterilní Milli-Q vody, tj. Vpož – Vpip (100 µl – 13,95 µl).
E) Otestování kvality DNA na agarosovém gelu
Na začátek >>